VA勉強会 #1
こんにちは、VA勉強会実行委員会です!
Vet Analyticsは獣医学に機械学習の知見を導入しようと日々奮闘しているデータサイエンス・エンジニアチームです。 東京大学本郷キャンパス付近でいつも活動しているので興味がありましたらtwitterアカウントから連絡してください。 Vet Analyticsは様々な方からのコンタクトを歓迎しています!
前回は交差検証法について解説しました!
今回は、VA勉強会で発表した論文について軽くまとめを書いていきます!
Deep Learningの動物行動学への応用事例について
今回発表した論文はこちらのレビューです
これは、行動学の研究者のツールの1つとしてDeep Learningがどのように使われているかをまとめた論文です。 (Open Accessなので是非読んでみてくださいね!)

動物行動学には上図のような技術が使われています。これらはDeep Learning分野でPose Estimation(姿勢推定)と呼ばれています。 特にDeepLabCutというツールが有名ですね。
筆者もこのツールを使って実験をしましたがなかなか制約条件が厳しくてうまくいかなかった思い出があります笑
本論文の筆者らは、Deep Learningを動物行動学に応用ためには以下の要素が必要だとしています
Can DNNs be harnessed with small training datasets? Due to the nature of ‘small-scale’ laboratory experiments, labeling 20, 000 or more frames is not a feasible approach (the typical human benchmark dataset sizes).
The end-result must be as accurate as a human manually-applied labels (i.e. the gold standard), and computationally tractable (fast).
The resulting networks should be robust to changes in experimental setups, and for long-term storage and re-analysis of video data, to video compression.
Animals move in 3D, thus having efficient solutions for 3D pose estimation would be highly valuable, especially in the context of studying motor learning and control.
Tracking multiple subjects and objects is important for many experiments studying social behaviors as well as for animal-object interactions.
1や2のようなデータセットの問題は多くのプロジェクトでボトルネックになることが多い障害の1つですね。
行動学は全く専門でないので5のような問題が重要になってくるのは意外でしたね。 特に獣医領域にDeep Learningを応用すると問題になるのが
データセットの不足(質・量共に)
エンジニア的リソースの不足
の2つだなという実感があります。しかし、2についてはGoogle Colabがだいぶ解消してくれているので実際は1番目の問題が多いように感じています。 本論文でもそのことについてがよく触れられていました。

上の図のように行動学は視覚的な評価に基づいていることが多いのでComputer Visionとの相性が良いことがわかりますね CV×獣医の研究がより出てくることを期待しています!
今回は以上です!ここまで読んでいただいてありがとうございました!
Vet Analyticsは様々な方からのコンタクトを歓迎しています!
ラットの疼痛評価方法への画像認識技術の応用
こんにちは、Vet Analyticsでデータアナリストを行っている富永です。
Vet Analyticsは獣医学に機械学習の知見を導入しようと日々奮闘しているデータサイエンス・エンジニアチームです。 東京大学本郷キャンパス付近でいつも活動しているので興味がありましたらtwitterアカウントから連絡してください。 Vet Analyticsは様々な方からのコンタクトを歓迎しています!
前回は交差検証法について解説しました!
今回は、獣医療への機械学習の応用事例についての論文について紹介したいと思います。
今回読んだ論文の概要
今回僕が紹介したいのは、「The Rat Grimace Scale: A Partially Automated Method for Quantifying Pain in the Laboratory Rat via Facial Expressions」と言う論文です。 www.ncbi.nlm.nih.gov
これは、機械学習の画像認識の技術を用いてラットの急性疼痛評価を半自動化しようとした論文です。
まずこの論文の先行研究には「Grimace Scale」と言うものがあります。 Grimace Scaleとは顔面の筋肉をいくつかのアクションユニット(AU)に分け、そのアクションユニットの動きを見ることで言葉を喋らない動物の疼痛を評価しようと言うものです。
この論文の先行研究として開発されたのがMGS(Mouse Grimace Scale)です。これは、マウスのための疼痛評価方法で「目、鼻、頬、耳、ひげ」の5つのポイントを見ています。 このポイントの評価を部分的に自動的にすることでこのスケールの有用性をあげようと言う狙いがあるみたいです。
この研究の一番大きな目標としては、「MGS(Mouse Grimace Scale)をRGS(Rat Grimace Scale)へ拡張させる」と言うことです。
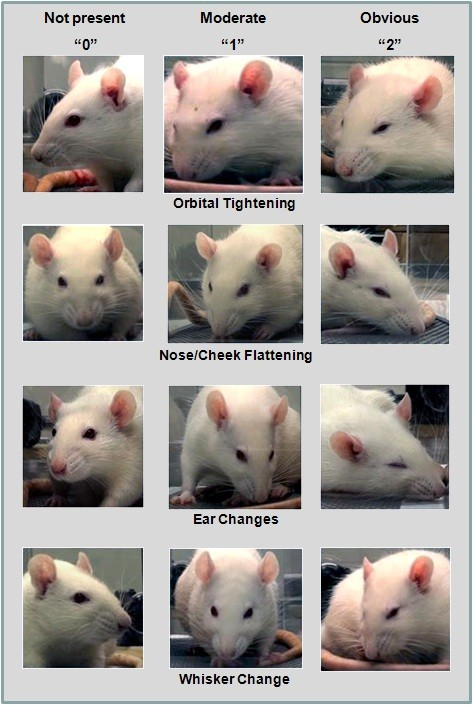
論文で使った手法について
この論文で僕が注目したポイントは3つ、「正解データとして用いる疼痛の評価方法は?」と「どのような機械学習モデルを使っているのか?」と「疼痛モデルの作成手法」です。
・正解データとして用いた疼痛評価方法
RGSによる疼痛評価と言うことはその評価が正しいかをジャッジする必要があると思います。そのジャッジする方法として、本論文では「CFAを注射した群を疼痛あり群」として用いています。5人のポスドク・学生がそれぞれ独立にスコアをつけ、その平均をペインスコアとして算出しているようです。その結果正解率は81.6%でありました。

・どのような機械学習モデルを使っているの?
機械学習モデルは、OpenCV2.0を使用しているみたいです。Open CVのHaar-Cascade classifierによってラットの顔と耳と目を矩形抽出しているみたいです。
そしてその抽出した矩形をパワポにランダムで貼り付けることで観察者の労働力を減らし評価へのバイアスをなくそうと言う試みのようです。


・疼痛モデルラットの作成
疼痛ありのラットの作成手としては、「ケイ酸アルミニウムとカラゲナン」もしくは「CFA」による炎症モデルと「卵巣摘出術を想定した回復手術」による周術期管理モデルの3つを使用しています。
このモデルでの疼痛評価も行っていて有意に疼痛の評価を行えています。

これらの結果からMGSが確実にRGSに拡張できていることがわかります!
論文についての自分の考え
この研究での機械学習の意味は「観察者の負担を減らす」と言う一点にのみフォーカスしていると言う意味で面白いなと感じました。最近の機械学習プロジェクトは、機械学習を前面に押し出しており Methodが新規性であることが多いように感じます。しかし、この研究のように研究のプロトコルの中で実験を円滑にするものとしての機械学習の使い方は、研究者を助けると言う意味での機械学習のあり方なのかなと思いました。このようなwet系の研究での機械学習の利用が促進されると面白いなと思いました。
次回は動的計画法(Dynamic Programing)について書きます。
Vet Analyticsは様々な方のコンタクトを歓迎しています!DMへのご連絡をお待ちしております!
交差検証を読み解く
こんにちは、Vet Analyticsでデータサイエンティストをしている浜野です。
Vet Analyticsは獣医学に機械学習の知見を導入しようと日々奮闘しているデータサイエンス・エンジニアチームです。 東京大学本郷キャンパス付近でいつも活動しているので興味がありましたらtwitterアカウントから連絡してください。
前回はロジスティック回帰の理論とPythonを使った実装方法について解説しました。
今回は交差検証について解説していきたいと思います。
はじめに
まず汎化誤差(test error)と訓練誤差(train error)について理解する必要があります。汎化誤差とは訓練データを使い統計手法で学習をした後、未知のデータに対して予測を行った場合の誤差です。また、訓練誤差は訓練に使ったデータに対して予測を行った場合の誤差です。
このときどのような基準でこの訓練誤差と汎化誤差を求めるのでしょうか。
バリデーションセット(交差)のアプローチ
このとき、バリデーションセットのアプローチでは、最も簡単なものとして、ランダムにデータを分割して、片方を訓練データ、もう片方をテストデータとします。 そしてそれぞれのデータに対して誤差を計算し、それを訓練誤差、汎化誤差とするパターンです。かなり直感的ですよね。
そこで実際にあるデータを使って実験してみました。
そしてそれぞれのデータに対して誤差を計算し、それを訓練誤差、汎化誤差とするパターンです。かなり直感的ですよね。
そこで実際にあるデータを使って実験してみました。
下の図は多項式回帰を使い、先ほど紹介したランダムに分割して求める手法を用いたものです。横軸は多項式回帰の次数、縦軸はMSEを用いた誤差です。

続いて、この試行を10回繰り返してみた結果がこちらです。一色が一回の試行を示しています。

こう見ると、一回の検証ごとの誤差にかなりばらつきがあることがわかりますね。どのデータが訓練に使われて、どのデータがテストに使われたか、というのがかなり誤差に響くことがわかりました。ここでこれらの解決策を2つ紹介します。
Leave-One-Out Cross-Validation
Leave-One-Out Cross-Validationは、「データを一つ取り出す(Leave-One-Out)」ことをし、それをテストデータ、それ以外を訓練データとする手法です。 この試行を全部がテストデータになる(つまりデータがn行ならn回)繰り返し、その平均をとり、それぞれを訓練誤差、汎化誤差としました。
しかしこの手法には欠点があります。計算量が莫大になってしまうことです。例えば1万行のデータセットがあったとしたら、1万回この試行を繰り返さなければならないわけです。これを改善したのが次の手法です。
この試行を全部がテストデータになる(つまりデータがn行ならn回)繰り返し、その平均をとり、それぞれを訓練誤差、汎化誤差としました。
しかしこの手法には欠点があります。計算量が莫大になってしまうことです。例えば1万行のデータセットがあったとしたら、1万回この試行を繰り返さなければならないわけです。これを改善したのが次の手法です。
K-fold Cross-Validation
K-fold Cross-Validationはデータをk個に均等に分割して、1つをテストデータ、それ以外を訓練データとする試行をk回繰り返し、平均をとる手法です。(注意:もしデータn行あったとしてn-fold Cross-Validationをしたとしたら、それはLeave-One-Out Cross-Validationと同じです)

この手法をLeave-One-Out Cross-Validationと比べてみましょう。
左がLeave-One-Out Cross-Validation、右が10-fold Cross-Validationを9回試した結果です。こうしてみてみると、10-fold Cross-Validationの方が計算量がずっと小さいのにもかかわらず、あまり結果が変わらないことがわかります。

実装
今回紹介したなかで最もよく使われるのは、やはりK-fold Cross-Validationです。簡単でライブラリも充実しています。
import numpy as np #こちらがK-fold Cross-Validationです from sklearn.model_selection import KFold X = np.array([[1, 2], [3, 4], [1, 2], [3, 4]]) y = np.array([1, 2, 3, 4]) #ここでfoldの数を指定します。今回は2とします kf = KFold(n_splits=2) KFold(n_splits=2, random_state=None, shuffle=False) for train_index, test_index in kf.split(X): print("TRAIN:", train_index, "TEST:", test_index) X_train, X_test = X[train_index], X[test_index] y_train, y_test = y[train_index], y[test_index]
出力結果
TRAIN: [2 3] TEST: [0 1] TRAIN: [0 1] TEST: [2 3]
しっかりK-fold Cross-Validationが実装できていますね! 以上で交差検証の説明は終わりです。 Vet Analyticsは様々な方のコンタクトを歓迎しています!DMへのご連絡をお待ちしております!
次回は趣向を変えて獣医学への機械学習の応用事例について書きたいと思います。
Vet Analyticsは様々な方のコンタクトを歓迎しています!DMへのご連絡をお待ちしております!
出典
とてもわかりやすい本です faculty.marshall.usc.edu
ロジスティック回帰の原理とPythonを用いた実装 ~基礎から実践まで~
こんには、Vet Analyticsでデータアナリストをやっている富永です。
Vet Analyticsは獣医学に機械学習の知見を導入しようと日々奮闘しているデータサイエンス・エンジニアチームです! 東京大学本郷キャンパス付近でいつも活動しているので興味がありましたらtwitterアカウントから連絡してください。
前回は機械学習の基本的なアルゴリズムの1つであるSVM(サポートベクトルマシン)について解説しました。 vetanalytics.hatenablog.com
今回は同じく基本的なアルゴリズムの1つであるロジスティック回帰(Logistic Regression)の原理とPythonによる実装について書いて行きます。
そもそもロジスティック回帰って??
ロジスティック回帰はよく研究に使われるので生物系研究者の方や獣医師の方にも馴染みが深いアルゴリズムの1つですね。 例えば、下のような研究でロジスティック回帰が用いられています。
これらの論文のようにロジスティック回帰を使っているを研究は数多く存在します。 しかし、なんとなく統計的処理の一環と思ってる方もいると思うのでこれを期に理解を深めましょう!笑
ロジスティック回帰は回帰という名前ながらも実際には分類問題に使われるアルゴリズムです。 基本的には二値分類(0 or 1のような)のためのモデルですが、拡張することで多クラスの分類に対しても 応用することができるようになります。
今回は、理論に関しては二値分類に関するアルゴリズムを実装に関しては多クラスの分類に関して扱っていきたいと思います!
ロジスティック回帰の理論
例えば疾患有りか疾患無しかを判定する問題があったとする。疾患有りの時の確率をpとすると、疾患無しの群の確率は1-pとなります。
よって疾患有りのオッズ比を見るととなります。オッズ比はその現象の起こりやすさを表す指標で、呼ばれるもので疫学研究などで出てくる指標の1つですね。今回はこの疾患の起こりやすさを表しています。
この対数をとったものをロジット関数といい、次の式になる。
この式は病気にかかっている確率を入れることでオッズ比の対数を出力すると言うものです。しかし、多くの問題の場合求めたいのは病気にかかっている確率ですよね。 なので、ロジット関数の逆関数をとると
となり、これによって出力が0から1までの確率になります!これをグラフ化すると次のようになります。

これのとき閾値関数を定めて二値分類問題として扱います。 この場合は、病気の確率が0.5以上のものを疾病群0.5未満のものをコントロール群とすると

これがロジスティック回帰による二値分類の基本的な仕組みです。
Pythonとscikit-learnを使ったロジスティック回帰の実装
今回も架空のデータを生成してロジスティック回帰による分類を行っていきたいと思います。
上記のような基礎的なロジスティック回帰では二値分類のみでしたが、Pythonモジュールの1つであるScikit-learnのロジスティック回帰では多クラス分類のモデル として拡張されています。
そこで今回は、scikit-learnを用いてロジスティック回帰で多クラス分類にチャレンジしたいと思います。
%matplotlib inline from sklearn.datasets import make_blobs import matplotlib.pyplot as plt from sklearn.linear_model import LogisticRegression as LR
まずは例の如く、scikit-learnと可視化のためのmatplotlibをインポートしていきましょう その次にロジスティック回帰を学習させるためのtrain dataとtest dataを作成していきましょう。
x_train,y_train = make_blobs(n_samples=200, #plotするサンプル数 n_features=2, #特徴量(2次元) centers=4, #クラスターの数 cluster_std=0.6, #クラスターごとの標準偏差 shuffle=True, random_state=0) x_test,y_test = make_blobs(n_samples=300, #plotするサンプル数 n_features=2, #特徴量(2次元) centers=4, #クラスターの数 cluster_std=0.8, #クラスターごとの標準偏差 shuffle=True, random_state=0)
これによってロジスティック回帰分類器を学習させる準備が整いました。 まずは、このサンプルの分散を確認してみましょう
plt.scatter(x_train[:,0], x_train[:,1], c="red", marker="o", s=40) plt.tight_layout() plt.show() plt.scatter(x_test[:,0], x_test[:,1], c="green", marker="o", s=40) plt.tight_layout() plt.show()


そして分類が確認できたら、学習と予測を行ってみましょう。
lr = LR(C=100.0, random_state=0) lr.fit(x_train, y_train) predict = lr.predict(x_test)
予測結果はpredictに格納されているので、これをplotしてみましょう。
plt.scatter(x_test[predict==0,0], x_test[predict==0,1], s=40, c="red", marker="o", label="1") plt.scatter(x_test[predict==1,0], x_test[predict==1,1], s=40, c="green", marker="o", label="2") plt.scatter(x_test[predict==2,0], x_test[predict==2,1], s=40, c="blue", marker="o", label="3") plt.scatter(x_test[predict==3,0], x_test[predict==3,1], s=40, c="yellow", marker="o", label="4") plt.legend(scatterpoints=1) plt.tight_layout() plt.show()

ちゃんとクラスターごとに分類できてることがわかりました。
次回は交差検証について書きます。
Vet Analyticsは様々な方のコンタクトを歓迎しています!DMへのご連絡をお待ちしております!
SVMの理論とPythonを使った実装方法について
こんにちは、Vet Analyticsでデータアナリストをしている富永です。
Vet Analyticsは獣医学に機械学習の知見を導入しようと日々奮闘しているデータサイエンス・エンジニアチームです! 東京大学本郷キャンパス付近でいつも活動しているので興味がありましたらtwitterアカウントから連絡してください。
前回はk-means法についての理論とpythonを使った実装方法について解説しました。 vetanalytics.hatenablog.com
今回はSVM(サポートベクトルマシン)の理論と実装方法について説明していきたいと思います!
SVM(サポートベクトルマシン)とは
SVM(サポートベクトルマシン)はかなりよく見る機械学習のアルゴリズムの1つで機械学習初心者ならば必ずおさえておきたい
アルゴリズムの1つです。
SVMは乱暴にいうと「クラスター同士の真ん中に境界を引くアルゴリズム」のことです。

上のように2つのクラスターがある時直感的には一本の境界が引けそうですよね?
でもその一本はどのようにして決めますか??

明確な定義がないと境界線が定まらないのです!
そこでSVMではこの境界の定義を
「その境界に一番近い点との距離の最大化」としました。

この境界線と直交するようなベクトル をwとすると この2つの点を通るような直線は
となるので、この2つの式から
これの両辺をベクトル の長さで割ると
となり、これは左辺が2つの境界線の距離を表しているので右辺を最大化すればいいことがわかりますね。 これは、
を最小化する問題と同値なので、 誤分類によるペナルティをpとすると 最小化すべき問題は
となります。(ここでCは定数) これを最小化することがSVMの1番の目標となるわけです。この問題をPythonを使って実装していきたいと思います。
SVMのpythonでの実装方法
今回はSVMをPythonモジュールのScikit-learnを用いて実装していきたいと思います。 まずは例によってサンプルデータの作成をしていきます。
%matplotlib inline from sklearn.datasets import make_blobs import matplotlib.pyplot as plt from sklearn.svm import SVC x_train,y_train = make_blobs(n_samples=200, #plotするサンプル数 n_features=2, #特徴量(2次元) centers=4, #クラスターの数 cluster_std=0.4, #クラスターごとの標準偏差 shuffle=True, random_state=0) x_test,y_test = make_blobs(n_samples=300, #plotするサンプル数 n_features=2, #特徴量(2次元) centers=4, #クラスターの数 cluster_std=0.8, #クラスターごとの標準偏差 shuffle=True, random_state=0)
この時にrandom_stateの値をtrainとtestで変えると予測モデルが使えなくなるので注意してください。 このコードによって生成されたサンプルデータをplotしていきます。
plt.scatter(x_train[:,0], x_train[:,1], c="red", marker="o", s=40) plt.tight_layout() plt.show() plt.scatter(x_test[:,0], x_test[:,1], c="green", marker="o", s=40) plt.tight_layout() plt.show()


次にtrainデータを用いて予測モデルの訓練と予測の実行を行っていきたいと思います。
svm = SVC(kernel="linear", C=1.0, random_state=0) svm.fit(x_train,y_train) predict = svm.predict(x_test)
この時Cがペナルティの許容度です。(前章参照) これで予測完了です。予測モデルの分類がうまくいっているかどうかを確かめてみましょう
plt.scatter(x_test[predict==0,0], x_test[predict==0,1], s=40, c="red", marker="o", label="1") plt.scatter(x_test[predict==1,0], x_test[predict==1,1], s=40, c="green", marker="o", label="2") plt.scatter(x_test[predict==2,0], x_test[predict==2,1], s=40, c="blue", marker="o", label="3") plt.scatter(x_test[predict==3,0], x_test[predict==3,1], s=40, c="yellow", marker="o", label="4") plt.legend(scatterpoints=1) plt.tight_layout() plt.show()

この予測はデータセットの形状からも納得できる結果だと思います。 しかし、訓練データと予測データの形状が異なる場合はどうなるのでしょうか?
SVMの予測失敗例
例えば、


のような直感的にもデータの分布が異なる場合について予測モデルを組み立てるとどうなるのでしょうか?
これに対して先ほどのように予測モデルを立てると

このように訓練データに対して予測データが異なると対応できないのが教師あり学習の弱いところです。 テストデータが学習時と似通っているほど精度が高くなります。
これは多くの機械学習プロジェクトで障壁となっている問題の1つです。 数多くのプロジェクトがこの障壁にぶつかり頓挫しました(笑) 次回以降にそれらのプロジェクトにも触れられたらと思います!
次回はロジスティック回帰について書きます。
Vet Analyticsは様々な方のコンタクトを歓迎しています!DMへのご連絡をお待ちしております!
K-means法の数式を読みとく
Vet Analyticsのデータサイエンティストをしてる浜野です。
Vet Analyticsは獣医学に機械学習の知見を導入しようと日々奮闘しているデータサイエンス・エンジニアチームです。 東京大学本郷キャンパス付近でいつも活動しているので興味がありましたらtwitterアカウントから連絡してください。
今回は機械学習で良く用いられるアルゴリズムの1つであるK-means法について紹介していきたいと思います。
数式
K-meansとは教師なし学習の階層クラスタリングという種類の機械学習の手法の一つになります。一言でいうと「決められたクラスタ数でデータを分割し、最適な分割法を探索する」という手法になります。下の図は150個の観測点に対し、違ったクラスタ数を指定しK-meansのアルゴリズムを適応した結果になります。

(An Introduction to Statistical Learning with Applications in R 著 Gareth James他)より
まずをそれぞれのクラスタの集合とします。この時K-meansアルゴリズムは下記の2つの条件を満たしている必要があります。
ここであるメジャー関数(コスト関数)をと定義します。この時、K-means法の問題は
を解く問題だと言い換えることができます。このメジャー関数の中の有名なものとしてはユークリッド距離があります。これは
と表すことができます。 これをわかりやすくいうと、k番目のクラスタのバリデーションは全てのクラスタ内の観測点のユークリッド距離の合計であり、これを最小化することが目的です。このメジャー関数を最初に式にいれると
となります。
アルゴリズム
数式を理解したところで実際にどのように実装するのか見ていきましょう
- まず観測点に対し、ランダムに1からKの番号をふっていく。これが初期状態のクラスタ配分となる。
- 全てのK個のクラスタに対し、重心を計算する。
- そしてそれぞれの観測点からどの重心が最も近いかを計算する(ここでユークリッド距離を使う)
- 2と3をk繰り返し、重心が動かなくなるまで続ける。
この手順を行うことで人気のアルゴリズムの1つであるk-means法を実装することができます。
scikit-learnを使って実際に実装してみる
今回はk-means法をpythonのモジュールの1つであるsikit-learnを使って実装していきたいと思います。 まず以下のように
%matplotlib inline from sklearn.datasets import make_blobs import matplotlib.pyplot as plt
において各種モジュール をインポートした後に(jupyter notebookを想定してます)
x,y = make_blobs(n_samples=200, #plotするサンプル数 n_features=2, #特徴量(2次元) centers=4, #クラスターの数 cluster_std=0.6, #クラスターごとの標準偏差 shuffle=True, random_state=0)
によってサンプル群を作成します。 本来k-means法は多次元に応用できるものですが、今回は二次元で行います。
plt.scatter(x[:,0], x[:,1], c="red", marker="o", s=40) plt.tight_layout() plt.show()

これでサンプル群が適切に生成されたのが確認できました! ここからが本題のk-means法による分割です。 scikit-learnのkmeansモジュール を使います。
from sklearn.cluster import KMeans kmeans_pre = KMeans(n_clusters=4, init="random", n_init=10, max_iter=300, tol=1e-04, random_state=0) p_km = kmeans_pre.fit_predict(x)
このコードでk-means法の推論が行われます。 k-means法は非常に計算効率が良いので高速で推論が終了します。
その結果を出力すると
plt.scatter(x[p_km==0,0], x[p_km==0,1], s=40, c="red", marker="o", label="1") plt.scatter(x[p_km==1,0], x[p_km==1,1], s=40, c="green", marker="o", label="2") plt.scatter(x[p_km==2,0], x[p_km==2,1], s=40, c="yellow", marker="o", label="3") plt.scatter(x[p_km==3,0], x[p_km==3,1], s=40, c="black", marker="o", label="4") plt.legend(scatterpoints=1) plt.tight_layout() plt.show()

正確にk-meansがクラスタ分類を行えたことがわかりました。
シュミレーション
このように実際にコードを動かす他にも 優れたシュミレーションがたくさんあるので、是非こちらのリンクで実際に試してみてください。
次回はSVM(サポートベクトル マシン)について書きます。
Vet Analyticsは様々な方のコンタクトを歓迎しています!DMへのご連絡をお待ちしております!